Artículos originales
Introducción del diagnóstico molecular de la hemocromatosis tipo 1 en Cuba
Introduction of Molecular Diagnosis of Hemochromatosis Type 1 in Cuba
Cómo citar este artículo:
Copyright: Esta revista provee acceso libre inmediato a su contenido bajo el principio de que hacer disponible gratuitamente investigación al publico apoya a un mayor intercambio de conocimiento global. Esto significa que se permite la copia y distribución de sus contenidos científicos por cualquier medio siempre que mantenga el reconocimiento de sus autores, no haga uso comercial de las obras y no se realicen modificaciones de ellas.

Recibido: 2012-06-12 21:42:53
Aprobado: 2013-01-07 11:25:19
Correspondencia: Ismael Aramís Cervera García. Facultad de Ciencias Médicas Finlay-Albarrán. La Habana. icervera@infomed.sld.cu
RESUMEN
Objetivo: establecer el diagnóstico de la hemocromatosis tipo 1 en Cuba, y calcular sus frecuencias en pacientes con hepatopatías.
Métodos: se realizó un estudio analítico y transversal conformado por 65 pacientes con hepatopatías, remitidos por genetistas clínicos al laboratorio de Biología Molecular del Centro Nacional de Genética Médica. Se empleó un PCR-RFLP para la detección de las mutaciones C282Y y H63D del gen HFE.
Resultados: se estandarizó el PCR-RFPL para la detección de las mutaciones C282Y y H63D. Las frecuencias de las mutaciones C282Y y H63D del gen HFE en los pacientes con afecciones hepáticas fueron de 6,3 % y 18,2 % respectivamente.
Conclusiones: el diagnóstico molecular de las mutaciones C282Y y H63D del gen HFE causantes de la hemocromatosis tipo 1 permitió identificar 28 portadores en los 65 pacientes estudiados, así como un individuo homocigoto para la mutación H63D, lo que expone la alta prevalencia de estas mutaciones en pacientes cubanos con hepatopatías.
Palabras clave: hemocromatosis; diagnóstico; técnicas de diagnóstico molecular; hepatopatías; cuba
ABSTRACT
Objective: to establish the diagnosis of hemochromatosis type 1 in Cuba, and calculate its frequencies in patients with hepatopathies.
Methods: an analytic cross-sectional study was conducted including 65 patients with liver disease, who were referred to the laboratory of Molecular Biology of the National Medical Genetics Center by clinical geneticists. A PCR-RFLP analysis was used for detecting the C282Y and H63D mutations in the HFE gene.
Results: PCR-RFLP analysis was standardized for the detection of C282Y and H63D mutations. Frequencies of C282Y and H63D mutations in the HFE gene in patients with hepatopathies were 6.3% and 18.2% respectively.
Conclusions: molecular diagnosis of C282Y and H63D mutations in the HFE gene causing hemochromatosis type 1 contributed to the identification of 28 carriers in the 65 patients who were studied, as well as a homozygous individual for the H63D mutation, which shows the high prevalence of these mutations in Cuban patients with liver disease.
Key words: hemochromatosis; diagnosis; molecular diagnostic techniques; liver diseases; cuba
INTRODUCCIÓN
La hemocromatosis tipo 1 es una enfermedad genética con herencia autosómica recesiva, caracterizada por una elevada absorción de hierro a nivel de las criptas intestinales como consecuencia de una modificación en su metabolismo, lo cual induce su depósito progresivo en distintos órganos, que da lugar al desarrollo de diferentes enfermedades.1,2
Esta hepatopatía es el defecto genético más frecuente en Occidente, donde afecta a 1 de cada 200 personas, principalmente de origen caucásico. 1,2 Se considera una enfermedad eventualmente grave debido a las lesiones celulares que pueden producirse en diferentes órganos y que durante gran parte de la vida puede desarrollarse de forma asintomática. Por esta razón, su diagnóstico pre sintomático (solo se pude realizar por estudios moleculares) y su posterior tratamiento con flebotomías, pueden impedir que la enfermedad devenga en: cirrosis, cardiopatías, diabetes mellitus y otras lesiones irreversibles.1,3
El gen cuyo defecto pude causar la hemocromatosis tipo 1 se denomina HFE y se encuentra en el brazo corto del cromosoma 6, en el locus 6p21,3, cerca del gen de HLA. Este gen codifica para una glicoproteína de 343 aminoácidos también denominada HFE, que interviene en la homeostasis del hierro a través de su unión al receptor de transferrina, que modula la interacción entre la transferrina y su receptor. 1,3,4 Las principales mutaciones del gen HFE que ocasionan esta hepatopatía son la sustitución de una cisteína por una tirosina en posición 282 de la proteína (C282Y), y el cambio de una histidina por un aspártico en la posición 63 (H63D). 1,4
Las técnicas que en la actualidad se utilizan para detectar la hemocromatosis tipo 1 consisten en la determinación del índice de saturación de transferrina (IST), el cual sitúa el umbral de sospecha por encima de valores del 45 %, la ferritina sérica (> 400 μg/L) y el hierro sérico, que debe ser mayor o igual a 150 μg/dL. Posteriormente se demuestra la presencia de mutaciones en el gen HFE por estudios moleculares. 3,5,6
El objetivo del presente estudio, por tanto, es el de instaurar el diagnóstico de la hemocromatosis tipo 1 en Cuba y establecer las prevalencias de las mutaciones C282Y y H63D en una muestra de pacientes con hepatopatías con el fin de obtener datos epidemiológicos que puedan permitir relacionar la presencia de estas mutaciones con la incidencia de la hemocromatosis tipo 1 en esta población.
MÉTODOS
Se realizó un estudio analítico y transversal para el universo de estudio conformado por los individuos no relacionados familiarmente, que presentaban antecedentes de hepatopatías crónicas, con marcadores virales e inmunológicos negativos, los cuales dieron su consentimiento para participar en esta investigación (en caso de los menores de edad, el consentimiento y la toma de la muestra la autorizó el tutor legal). Dicho universo de estudio estuvo constituido por el ADN extraído de 65 pacientes con hepatopatías procedentes de distintas provincias de Cuba, remitidos por los genetistas clínicos al laboratorio de Biología Molecular del Centro Nacional de Genética Médica.
El ADN se obtuvo a partir de una muestra 10mL de sangre periférica, recolectadas en un tubo estéril, con el anticoagulante etilendiaminotetraacetato (EDTA) a una concentración de 56mg/mL. La extracción de ADN fue realizada por el método de precipitación salina, descrito por Miller SA, en 1986.7
Después de procesar las muestras a través de un código se extrajo el ADN y se almacenaron en un banco de ADN con condiciones óptimas de temperatura y seguridad y solo fueron utilizadas con la finalidad de desarrollar esta investigación.
Para la amplificación de las regiones de interés del gen HFE, ubicados en los exones 2 y 4 del gen HFE, fue utilizada la técnica de PCR.
Este PCR consistió en la amplificación de las regiones flanqueadas por los cebadores HLAH-4 y HLAH-5. 8 (Tabla 1).
La amplificación fue llevada a cabo en un volumen final de 25ųL que contiene 100ng de ácido desoxirribonucleico (ADN) genómico, 0,2ųM del cebador HLAH-4, 0,2ųM del cebador HLAH-5, 1X del tampón suministrado con la enzima, 1,5mM de MgCl2, 0.25mM de 2’- desoxinucleosido 5’-trifosfato (dNTP) y 1,5U Taq ADN Polimerasa (Invitrogen).
El proceso de amplificación se realizó en el termociclador MJ Research, Estados Unidos. En el que se programó una desnaturalización inicial del ADN a 95ºC por tres minutos. La amplificación incluyó 30 ciclos de desnaturalización/ hibridación/ extensión: 60 segundos a 94ºC, 60 segundos a 54ºC, que constituye la TH de los cebadores utilizados y 60 segundos a 72ºC, respectivamente. La amplificación concluyó con 10 minutos a 72ºC para la extensión final.
Amplificación del exón 4 del gen HFE
El montaje del PCR para la amplificación de las regiones flanqueadas por los cebadores HLAH-1 y HLAH-2. 8 contiene: 100ng de ADN genómico, 0,2ųM del cebador HLAH-1, 0,2ųM del cebador HLAH-2, 1X del tampón suministrado con la enzima, 1,5mM de MgCl2, 0,25mM de dNTP y 1,5U Taq DNA Polimerasa (Invitrogen), en un volumen final de 25ųL. (Tabla 2).
El proceso de amplificación se realizó en el termociclador MJ Research, EE.UU, donde se desarrolló una desnaturalización inicial del ADN a 95ºC por tres minutos. La amplificación incluyó 30 ciclos de los pasos de desnaturalización/ hibridación/ extensión de 60 segundos a 94ºC, 60 segundos a 60ºC, y 60 segundos a 72ºC, respectivamente.
La amplificación concluyó con 10 minutos a 72ºC, para la extensión final.
Digestión enzimática
Para la determinación de la presencia o no de las mutaciones más frecuentes del gen HFE en las regiones amplificadas por la técnica de PCR, fueron realizadas digestiones enzimáticas con enzimas de restricción específicas en cada uno de los casos. 8
Digestión enzimática para el producto de la PCR del exón 2
La digestión enzimática fue llevada a cabo en un volumen final de 20ųL que contiene 8ųl de producto de PCR, 1X del tampón suministrado con la enzima y 10U de la enzima de restricción Mbo I (Invitrogen). Esta mezcla se incubó a 37ºC toda la noche.
Digestión enzimática para el producto de la PCR del exón 4
La digestión enzimática fue llevada a cabo en un volumen final de 20ųL que contiene 8ųl de producto de PCR, 1X del tampón suministrado con la enzima, 80ng/ųL de albúmina de suero bobino (BSA) y 30U de la enzima de restricción Rsa I (Promega). Esta mezcla se incubó a 37ºC toda la noche.
Electroforesis en gel de agarosa
Los productos de PCR fueron sometidos a electroforesis en gel de agarosa al 2 %, teñido con bromuro de etidio 5X. Se mezclaron 5ųl de cada producto amplificado, así como de los controles y/o patrón de peso molecular, con 1ųl de bromofenol azul (BFA) como frente de corrida. Se utilizó el tampón TBE 0,5X (0,5M de Tris, 10mM de EDTA y 0,5mM de ácido bórico) para cada corrida electroforética y se aplicó un voltaje de 250V, por 30 minutos. El producto amplificado se visualizó por exposición a la luz ultravioleta en un transluminador.
Interpretación de los resultados
PCR y digestión enzimática del exón 2.
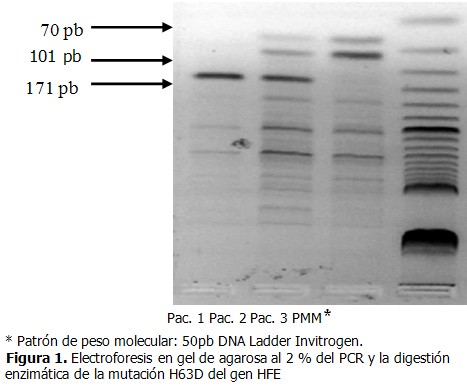
- El producto de PCR del exón 2 corresponde a 171 pares de bases (pb). La digestión enzimática con la enzima Mbo I genera dos fragmentos de 101pb y 70pb que corresponden al alelo normal.
- En el alelo patológico desaparece el sitio de restricción, y se obtiene el fragmento íntegro de 171pb.
PCR y digestión enzimática del exón 4.
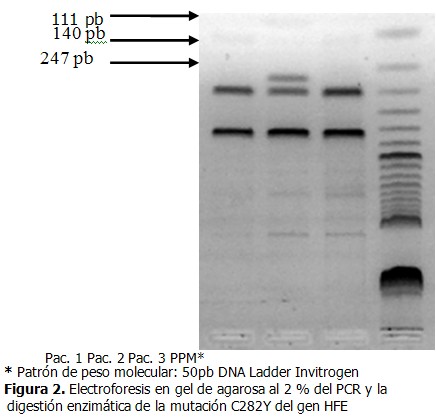
- El producto de PCR del exón 4 corresponde a un fragmento de 387pb. El producto del PCR digerido con la enzima Rsa I del alelo normal genera dos fragmentos de 247pb y 140pb.
- En el alelo mutado se genera un nuevo sitio de restricción, y se obtienen tres fragmentos de 247pb, 111pb y 29pb.
RESULTADOS
Estandarización de la técnica de PCR-RFLP para la detección de la mutación H63D
La estandarización del PCR de la mutación H63D se efectuó como se describe anteriormente. Esta mutación consistió en un cambio de G por A en el nucleótido 187 en el gen HFE, que ocasionó que desapareciera el sitio de restricción de la enzima Mbo I localizado en el exón 2, como consecuencia se obtiene un fragmento de 171pb. 8
Se muestra la corrida electroforética de tres pacientes diagnosticados clínicamente con hemocromatosis tipo 1 a los que se les realizó el PCR para la detección de la mutación H63D y un marcador de peso molecular de 50pb. La carrilera uno se correspondió con el paciente uno, en este caso se obtuvo un fragmento de 171pb que se correspondió con el alelo patológico, por lo que desapareció el sitio de restricción y resultó homocigoto para la mutación H63D. En la carrilera dos se pudieron apreciar tres fragmentos de 171pb, 101pb y 70pb los que correspondieron a los alelos normal y mutado por lo que se puede decir que este paciente dos, resulta heterocigótico para la mutación H63D. En la carrilera tres, correspondiente al paciente tres, se obtuvieron dos fragmentos, de 101pb y 70pb que se correspondieron al alelo normal, por tanto este paciente no presentó dicha mutación. (Figura 1).
Estandarización de la técnica de PCR-RFLP para la detección de la mutación C282Y
La estandarización del PCR de la mutación C282Y se efectuó como se describió anteriormente. Esta mutación, que consiste en un cambio de G por A en el nucleótido 845 del gen HFE crea un segundo sitio de restricción en el exón 4 para la enzima Rsa I, como resultado se obtienen tres fragmentos de 147pb, 11pb y 29pb. 8
La figura se corresponde a una corrida electroforética de las muestras de tres pacientes diagnosticados clínicamente con hemocromatosis tipo 1, a los que se les realizó el PCR y su posterior digestión enzimática con la enzima Rsa I para la detección de la mutación C282Y, y un marcador de peso molecular de 50pb. En los casos uno y tres se obtuvieron dos fragmentos de 247pb y 140pb que corresponden con el alelo normal, es decir estos pacientes no presentaron la mutación C282Y y en el caso dos se obtuvieron tres fragmentos de 247pb, 140pb y 111pb que se relacionan con el alelos normal y el patológico, por lo que este paciente resulta heterocigótico para dicha mutación. No se logró distinguir el fragmento de 27pb debido a que la electroforesis se realizó en un gel de agarosa al 2 %. (Figura 2).
Mutación C282Y del gen HFE en los pacientes con afecciones hepáticas
De los 65 individuos analizados, siete resultaron ser heterocigóticos para la mutación C282Y del gen HFE, mientras que el resto no presentaron la mutación. No se detectaron individuos homocigóticos. Estos resultados muestraron una frecuencia del alelo mutado de 0,053 (5,3 %) en esta población estudiada.
Mutación H63D en los pacientes estudiados con hepatopatías
Se realizó la detección de la mutación H63D del gen HFE en 65 pacientes con hepatopatías. De los 65 individuos analizados uno resultó ser homocigótico para la mutación H63D del gen HFE, 21 fueron heterocigóticos, y el resto no presentaron esta mutación. Estos resultados proporcionaron una frecuencia del alelo mutado de 0,17 (17 %) en la población estudiada.
DISCUSIÓN
Mutación C282Y del gen HFE en los pacientes con afecciones hepáticas
La prevalencia de la mutación C282Y ha sido muy estudiada en diferentes poblaciones europeas.9 Algunos estudios han postulado el origen celta de esta mutación, que refleja un gradiente descendiente con una incidencia de norte a sur, acentuada en su mayor frecuencia en poblaciones del norte de Europa: Finlandia (5,2 %), Gran Bretaña (5,9 - 8,5 %), Islandia (6,7 %), Noruega (6,4 %) o Irlanda (10 %). Por el contrario, la menor prevalencia se encuentra en el área mediterránea, con frecuencias del 1,4 % en Grecia, el 1,3 % en Italia y del 0 % en Turquía. En la población española, la prevalencia de esta mutación se sitúa en valores comprendidos entre el 4,4 % en Cantabria y el 2 % en la región central. Otros estudios han estimado una frecuencia para la mutación C282Y del 3 % y del 3,6 % en población catalana y vasca, respectivamente, aunque con un número de individuos analizados tan reducido que pueda haber dado lugar a una sobreestimación de esta prevalencia. Otro trabajo realizado con población del País Vasco sitúa esta prevalencia en un 5,2 %. 4,9
Los resultados obtenidos en este grupo de pacientes con una prevalencia de la mutación C282Y del gen HFE del 5,3 %, están dentro de los valores estimados para la población española, debido posiblemente a que la población cubana actual es el resultado de una contribución mixta entre la población española y la del África Subsahariana principalmente.
Mutación H63D en los pacientes estudiados con hepatopatías
Desde su descubrimiento la mutación H63D ha suscitado controversia sobre su confirmación como tal o su consideración como un mero polimorfismo, debido a que la mutación se encuentra presente en la población Europea con una frecuencia relativamente alta (media de 14 %) y en aquellas de descendencia europea, lo que está en correspondencia con los resultados obtenidos en este trabajo. En el norte de África, en el Medio Este, y en partes de Asia su frecuencia es moderada. 9
Conjuntamente numerosos autores han planteado que en todas las poblaciones europeas y en aquellas de descendencia europea, la mutación H63D es más frecuente que la C282Y, resultados que son comparables con los hallados en este estudio. 1,9
Al mismo tiempo, y teniendo en cuenta que la mutación H63D potencialmente provoca un aumento en la absorción del hierro, aunque menor que el provocado por la mutación C282Y, pudiera ser que en algunos de los pacientes estudiados esta mutación incidiera o incluso fuera la causante de la afección hepática de estos al interactuar con otros genes o con el medio ambiente en que se desenvuelve el paciente, tal como se ha descrito en el caso de la mutación C282Y.
El diagnóstico molecular de las mutaciones C282Y y H63D del gen HFE causantes de la hemocromatosis tipo 1 permitió identificar 28 portadores en los 65 pacientes estudiados, así como un individuo homocigoto para la mutación H63D, lo que expone la alta prevalencia de estas mutaciones en pacientes cubanos con hepatopatías.
REFERENCIAS BIBLIOGRÁFICAS
- Cervera IA. Hemocromatosis tipo I. Patogenia y diagnóstico. Medisur. 2012;10(2):1-20 [Buscar en Google Scholar]
- Adams PC, Barton JC. Haemochromatosis. The Lancet. 2007;370(9602):1855-60 [Buscar en Google Scholar]
- Pietrangelo A. Hereditary hemochromatosis:pathogenesis, diagnosis and treatment. Gastroenterology. 2010;139(2):393-408 [Buscar en Google Scholar]
- Torremocha RA, Salido MC, Santiago C, Chicharro L, López I, Martín MJ. Prevalencia de mutaciones C282Y, H63D y S65C en un colectivo laboral del País Vasco. Rev Diagn Biol. 2002;51(4):140-4 [Buscar en Google Scholar]
- Bacon B, Adams P, Kowdley K, Powell L, Tavill A. Diagnosis and management of hemochromatosis:2011 practice guideline by the American Association for the Study of Liver Diseases. Hepatology. 2011;54(1):328-43 [Buscar en Google Scholar]
- Jouanolle AM, Gérolami V, Ged C, Grandchamp B, Le Gac G, Pissard S, et al. Molecular diagnosis of HFE mutations in routine laboratories. Results of a survey from reference laboratories in France. Ann Biol Clin (Paris). 2012;70(3):305-13 [Buscar en Google Scholar]
- Miller SA, Dykes DD, Polesky HF. A simple salting out procedure for extracting DNA from human nucleated cells. Nucleic Acids Res. 1988;16(3):1215 [Buscar en Google Scholar]
- Feder JN, Gnirke A, Thomas W, Tsuchihashi Z, Ruddy DA, Basava A, et al. A novel MHC class I-like gene is mutated in patients with hereditary haemochromatosis. Nat Genet. 1996;13(4):399-408 [Buscar en Google Scholar]
- Sassi R, Hmida S, Kaabi H, Hajjej A, Abid A, Abdelkefi S, et all. Prevalence of C282Y and H63D mutations in the haemochromatosis (HFE) gene in Tunisian population. Ann Genet. 2004;47(4):325-30 [Buscar en Google Scholar]
Enlaces refback
- No hay ningún enlace refback.
FINLAY EN: 








FINLAY CERTIFICADA POR:

Esta revista "no aplica" cargos por publicación en ninguna etapa del proceso editorial.
Dirección postal: Calle 51A y Avenida 5 de Septiembre Cienfuegos, Cuba Código postal: 55100.
http://www.revfinlay.sld.cu
Telefono: +53 43 516602. Telefax: +53 43 517733.
amgiraldoni@infomed.sld.cu
ISSN: 2221-2434
RNPS: 5129